 Comment les 2 mètres d'ADN qui se trouvent dans le noyau de chacune de nos cellules sont ils repliés ? Comment cette organisation permet-elle le fonctionnement correct de nos chromosomes ? Pour répondre à ces questions, des biologistes moléculaires ont mis au point dans les années 2000 la technique de Capture de Conformation des Chromosomes (3C). Elle consiste à fixer chimiquement les chromosomes, à couper la molécule d'ADN en petits fragments puis à relier entre eux les fragments qui se trouvent à proximité. Ces fragments sont ensuite séquencés, ce qui permet de dresser une carte de contacts du génome complet (voir figure) ; carte qui reflète ainsi le repliement des chromosomes. Mais comment lire cette carte ?
Comment les 2 mètres d'ADN qui se trouvent dans le noyau de chacune de nos cellules sont ils repliés ? Comment cette organisation permet-elle le fonctionnement correct de nos chromosomes ? Pour répondre à ces questions, des biologistes moléculaires ont mis au point dans les années 2000 la technique de Capture de Conformation des Chromosomes (3C). Elle consiste à fixer chimiquement les chromosomes, à couper la molécule d'ADN en petits fragments puis à relier entre eux les fragments qui se trouvent à proximité. Ces fragments sont ensuite séquencés, ce qui permet de dresser une carte de contacts du génome complet (voir figure) ; carte qui reflète ainsi le repliement des chromosomes. Mais comment lire cette carte ?
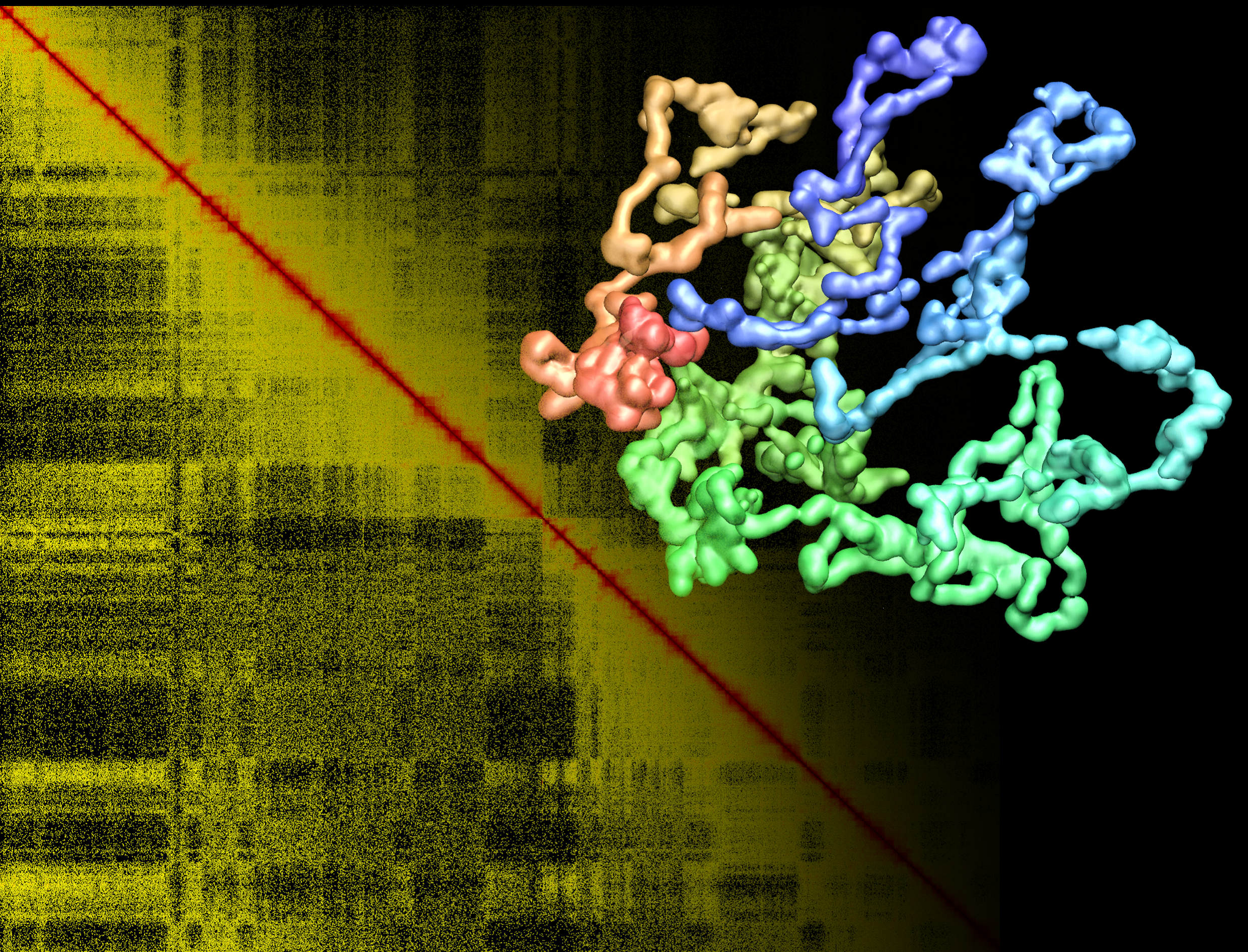
Pour exploiter au mieux les données contenues dans de telles cartes, Lesne at al. ont mis au point un algorithme capable de reconstruire la structure 3D des chromosomes à partir des points de contacts déterminés expérimentalement. Son principe est d'utiliser la carte de contacts pour engendrer un graphe (deux sites seront reliés par un lien s'ils sont en contact) puis d'estimer la distance relative entre deux sites génomiques quelconques comme le nombre minimal de liens nécessaires pour passer de l'un à l'autre sur le graphe. Des méthodes mathématiques permettent alors d'estimer les coordonnées tri-dimensionnelles des sites à partir de la connaissance de leurs distances
La structure obtenue peut ensuite être croisée avec d'autres jeux de données génomiques, comme par exemple la position de certaines protéines le long du génome pour visualiser les structures formées en trois dimensions par ces protéines dans le noyau ! Cet outil pourrait bientôt permettre la réalisation d'un navigateur 3D qui permettrait d'étudier notre génome à la manière dont Google Earth nous permet de survoler virtuellement la planète.

Figure: le fond montre une carte de contacts chromosomiques humains après traitement et normalisation des données expérimentales obtenues in vivo: plus un pixel est de couleur rouge, plus la fréquence de contact entre les deux sites génomiques associés (en abscisse et en ordonnée) est élevée. L'algorithme proposé par Lesne et al. permet de reconstruire la structure 3D correspondante, ici le chromosome 1 où le gradient de couleur indique la position le long du génome.
En savoir plus:
3D genome reconstruction from chromosomal contacts, Annick Lesne, Julien Riposo, Paul Roger, Axel Cournac, Julien Mozziconacci, Nature Methods (2014) doi: 10.1038/nmeth.3104,publication en ligne le 21 septembre 2014.
http://www.nature.com/nmeth/journal/vaop/ncurrent/full/nmeth.3104.html
Contact:
Annick Lesne, CNRS <
Julien Mozziconacci, UPMC-Paris 6




